
合成生物学聚焦于各种生命系统工程化和模块化再造,并已经在能源,医药等领域取得了一些显著性成果。丝状微生物(真菌及放线菌等)是次级代谢产物的主要来源,约有90%的已知抗生素由这类微生物产生,其中三分之二以上是以链霉菌为代表的放线菌产生的。原核的链霉菌基因组比真核的酵母菌还要大,基因还要多,其中三分之二的基因参与调控过程。然而由于链霉菌丝状生长的特性使其基因表达调控难以精确设计、定量和预测,成为合成生物学在该领域发展的一个巨大的技术瓶颈。由张立新研究员担任首席科学家的973项目“合成微生物体系的适配性研究”正是试图解决这一科学问题,提升对链霉菌生命规律的认识层次,实现对链霉菌生命过程的定量预测、精准化设计、标准化合成与精确调控。项目自2013年实施以来,团队成员坚持每个月开例会协同攻关,促进各课题组的交叉融合。仅微生物所参加的谭华荣、杨克迁、陶勇、唐双燕、陈义华、娄春波等课题组就在Nature及其子刊、PNAS、Angew、MM、ME等顶尖杂志上发表了合作成果。
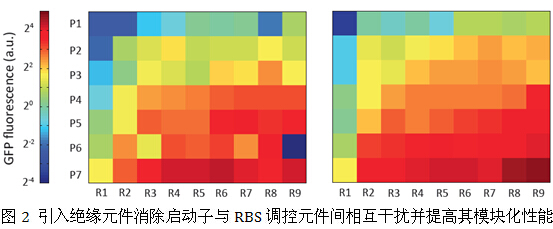
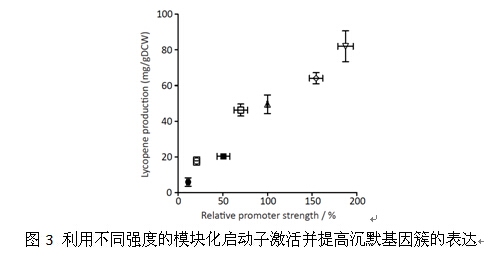
最近,娄春波课题组和张立新课题组合作首次针对链霉菌生物元件建立了基于流式细胞仪和报告基因(sfGFP)的单细胞精确定量方法。该方法通过优化原生质体制备方案,替换了对流式细胞仪有损害的蔗糖缓冲体系,同时结合PI染色以分离出死细胞个体,解决了丝状微生物生长形态分化和程序性死亡不利于定量的缺陷,从而提高了测量精度。在高通量地刻画了覆盖范围足够大的启动子和RBS等调控元件后,又成功地利用RiboJ绝缘子在链霉菌中消除了启动子和RBS的干涉效应,提高了这些元件模块化性能。这些模块化的调控元件也被成功地应用于激活阿维链霉菌中沉默基因簇番茄红素的表达,并通过可预测地替换调控元件大大提高了番茄红素的产量,达到工业生产的要求。该研究工作为应用合成生物学理念改造丝状微生物次级代谢产物及激活沉默基因簇奠定了重要基础。
上述研究结果发表在美国《国家科学院院刊》上。张立新课题组白超弦及娄春波课题组博士生张洋,助理研究员赵学金为论文的共同第一作者。张立新研究员和娄春波研究员为通讯作者,其他作者还有胡逸灵、向四海博士和苗靳博士。该研究得到了“973”项目和国家自然科学基金项目的资助。(来源:科学网)