|
|
|
|
|
组蛋白修饰可调控mRNA剪切来决定胚胎干细胞命运 | Genome Biology |
|
|
论文标题:Alternative splicing links histone modifications to stem cell fate decision
期刊:Genome Biology
作者:Yungang Xu†, Weiling Zhao†, Scott D. Olson, Karthik S. Prabhakara and Xiaobo Zhou
发表时间:2018/09/14
数字识别码:10.1186/s13059-018-1512-3
原文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-018-1512-3?utm_source=WeChat&utm_medium=Website_linksSocial_media_organic&utm_content=CelZha-MixedBrand-multijournal-Multidisciplinary-China&utm_campaign=ORG_AWA_CZH_BMCWechat_dailyposts_blogs
微信链接:https://mp.weixin.qq.com/s/ur38n0xcRb_xInnaG4ej2Q
胚胎干细胞(ESC)具有体外培养无限增殖、自我更新和多向分化的特性。解析ESC细胞定向分化决定机制对于发育生物学和再生医学至关重要。已有研究表明可变剪切、细胞周期控制和组蛋白修饰等在ESC的定向分化中起着重要的作用。然而,这些机制之间的复杂关联以及它们如何共同作用并参与ESC的命运决定还有待研究。
美国德克萨斯大学医学中心(https://sbmi.uth.edu/ccsm/)周小波教授领导的研究团队9月14日在Genome Biology发表最新研究成果,揭示了组蛋白修饰可通过调控细胞周期相关转录因子或通路基因的可变剪切进而参与ESC定向分化的调控。周小波教授为论文的通讯作者,徐云刚助理教授和赵蔚苓副教授为共同第一作者;该校McGovern医学院Scott D. Olson助理教授和 Karthik S. Prabhakara博士为共同作者。
本文研究者用人类胚胎干细胞(hESC)H1细胞系以及由其诱导分化而来的四个细胞类型(包括中胚层细胞ME、滋养层类细胞TBL、神经母细胞NPC和间质干细胞MSC)来代表五个不同分化程度的细胞谱系(cell lineage),并用IMR90用作终端分化体细胞的参照细胞。作者对这些细胞的转录组和表观组进行了整合分析(图1)。通过对转录组分析, 研究者首先鉴定了数千个与hESC分化关联的可变剪切事件,这些事件的剪切模式很好的表征了这些不同细胞谱系的分化程度和组织关系。

图1. 本文研究的hESC分化示意图及其组织学关系(原文Figure S1A)
先前在单一基因水平上的研究表明,组蛋白修饰可以参与调控转录耦合的可变剪切事件[1, 2],进而揭示了组蛋白修饰不但可以决定基因的表达水平(比如通过启动子和增强子表观修饰),还可以决定基因是如何剪接的。然而,尚不清楚组蛋白修饰和可变剪切在全基因组水平上的关联以及它们是如何共同参与某一生物过程的(如细胞分化)。因此,本文研究者整合分析了上述6种细胞的16种组蛋白修饰(包括9种组蛋白乙酰化修饰和7种组蛋白甲基化修饰)和转录组。研究者发现在hESC分化前后,绝大多数组蛋白修饰在可变剪切的外显子周围的动态变化要显著于其它组成型外显子(图2A),表明组蛋白修饰可能与可变剪切调控有关。进一步的关联分析表明,其中3种组蛋白修饰(H3K36me3、H3K27ac和H4K8ac)与大约52%的可变剪切事件有显著的正或负相关性(图2B)。

图2. 组蛋白修饰与可变剪切关联分析。A. 组蛋白修饰的动态变化主要发生在可变剪切的外显子周围。B. H3K27ac及其显著正相关的可变剪切事件。(原文Figure 3A和3D)
作者进一步分析了与组蛋白修饰紧密关联的可变剪切基因的功能,以期揭示它们在ESC定向分化中的作用。分析表明,与组蛋白修饰关联的可变剪切基因与干细胞的干性密切相关(图3A),且主要受到细胞分化相关转录因子的调控(图3B)。进一步的功能富集分析表明组蛋白修饰关联的可变剪切基因主要参与G2/M期过程以及DNA损伤修复,而其它可变剪切基因主要与G1/S期过程和Wnt通路有关(图3C)。而之前已有研究表明在细胞分化过程中,细胞周期对于细胞命运的决定起了至关重要的作用。其中G1/S期是细胞接收外部刺激获得分化指令的阶段; 而G2/M期是细胞执行分化指令并最终决定细胞分裂后及其命运的阶段[3-5]。本文的研究将组蛋白修饰、可变剪切和细胞周期相关的机制关联起来,首次提供了一种胚胎干细胞定向分化的综合调控机制。

图3. 组蛋白修饰关联的可变剪切基因与其它基因具有显著不同的功能富集。A. 组蛋白修饰关联的可变剪切基因与干细胞密切相关;B. 组蛋白修饰关联的可变剪切基因主要受控于细胞分化有的关转录因子;C. 组蛋白修饰关联的可变剪切基因与其它可变剪切基因分别富集于不同的细胞周期及相关通路。(原文Figure 4A-C)
最后,作者通过对ENCODE/Roadmap数据的分析和实验验证,证实了H3K36me3可以通过PSIP1/SRSF1适配器复合体来调节PBX1的可变剪切,进而决定hESC的分化命运。PBX1蛋白是TALE家族的同源异型结构域(homeodomain)转录因子。其主要功能涉及淋巴母细胞白血病[6]和多种癌症[7-12],调控发育相关基因表达[13],以及参与细胞周期调控[14]。PBX1基因有三个转录同源体 (RefSeq注释),PBX1a、PBX1b和PBX1c。其中PBX1a和PBX1b是主要的转录本,由第7个外显子的可变剪切产生。本文研究者发现,PBX1a和PBX1b的表达在hESC分化过程中与第7个外显子周围的H3K36me36修饰强度显著正相关;对ENCODE/Roadmap收录的56种细胞和组织的数据进行分析也进一步证实了此相关性。PBX1a主要在干细胞中高表达,可通过调节NANOG基因表达进而激活多能性转录调控网络来促进干细胞增殖(图4);尽管具有与PBX1a相同的DNA结合结构域,但PBX1b在已分化细胞中高表达。由于PBX1b缺失了C末端的功能结构域,进而推测其丧失了PBX1a的转录调控活性,进而抑制了多能性转录调控网络在已分化细胞中的活性。为了证实该推测,本文作者进一步对H1细胞系、由H1诱导分化而来的MSC细胞以及来自病人的两株MSC细胞株系和IMR90细胞进行了大量的分子生物学实验分析(ChIP-PCR, RIP-PCR, western-blot等)分子实验,证实了H3K36me3在H1细胞和其它细胞间的变化以及PSIP1和SRSF1分别在染色质和pre-mRNA的第7外显子周围结合的变化。以上结果表明,PBX1基因的可变剪切可将H3K36me3修饰与ESC细胞分化的命运关联起来(图4),进而例证了组蛋白修饰可调控mRNA剪切来决定ESC细胞命运。
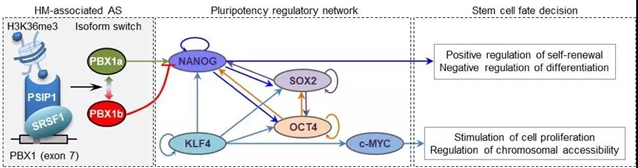
图4. PBX1基因可变剪切将H3K36me3与hESC分化连接起来 (原文Figure 6F)
主要作者简介

通讯作者:周小波
美国德州大学医学中心终身正教授、Dr. & Mrs. Carl V. Vartian教授、计算与系统医学研究中心主任。主要从事数据挖掘、机器学习、生物信息学、系统生物学、生物医学成像、再生医学、临床与转化医学信息学、与手术设计与优化等的研究。先后在北大、清华、兰大、华为、Missouri-Colombia大学、Texas A&M大学、Harvard大学、Cornell大学、维克森林大学、德州大学医学中心从事大数据,生物医学信息学与影像等的研发工作。

第一作者:徐云刚
美国德州大学医学中心助理教授。2014年获得哈尔滨工业大学生物信息学博士学位,同时具有生物学学士和硕士学位。先后在维克森林大学和德州大学医学中心从事博士后研究工作,主要研究方向为生物信息学和计算生物学。具体研究内容包括基因和miRNA双层网络重构、建模与分析等系统生物学领域;SNP、GWAS、NGS数据处理与分析等功能基因组学领域;以及算法设计、统计模型构建、深度学习方法开发等数据挖掘领域。目前主要从事基于高通量测序数据(含单细胞测序)的表观组和转录组调控研究。

共同第一作者:赵蔚苓
美国德州大学医学中心副教授,分别在1998年和2000年获得爱荷华大学医学硕士和博士学位。先后在爱荷华大学,维克森林大学和德州大学医学中心从事放射医学和生物信息医学相关的研究。她在放射医学,氧化应激诱导和癌症生物学有着多年的丰富研究经验。
参考文献
1. Luco RF, Pan Q, Tominaga K, Blencowe BJ, Pereira-Smith OM, Misteli T: Regulation of alternative splicing by histone modifications. Science 2010, 327:996-1000.
2. Luco RF, Allo M, Schor IE, Kornblihtt AR, Misteli T: Epigenetics in Alternative Pre-mRNA Splicing.Cell 2011, 144:16-26.
3. Pauklin S, Vallier L: The Cell-Cycle State of Stem Cells Determines Cell Fate Propensity (vol 155, pg 135, 2013). Cell 2014, 156:1338-1338.
4. Vallier L: Cell Cycle Rules Pluripotency. Cell Stem Cell 2015, 17:131-132.
5. Dalton S: Linking the Cell Cycle to Cell Fate Decisions. Trends in Cell Biology 2015, 25:592-600.
6. Foa R, Vitale A, Cuneo A, Mecucci C, Mancini H, Cimino G, Cutinelli F, Saglio G, Torelli G, Quarta G, et al: E2A-PBX1 fusion in adult acute lymphoblastic leukemia (ALL) with t(1;19) translocation: Biologic and clinical features. Blood 2000, 96:189b-189b.
7. Jung JG, Kim TH, Gerry E, Kuan JC, Ayhan A, Davidson B, Shih IM, Wang TL: PBX1, a transcriptional regulator, promotes stemness and chemoresistance in ovarian cancer. Clinical Cancer Research2016, 22.
8. Magnani L, Patten DK, Nguyen VTM, Hong SP, Steel JH, Patel N, Lombardo Y, Faronato M, Gomes AR, Woodley L, et al: The pioneer factor PBX1 is a novel driver of metastatic progression in ER-positive breast cancer. Oncotarget 2015, 6:21878-21891.
9. Feng Y, Li L, Zhang X, Zhang Y, Liang Y, Lv J, Fan Z, Guo J, Hong T, Ji B, et al: Hematopoietic pre-B cell leukemia transcription factor interacting protein is overexpressed in gastric cancer and promotes gastric cancer cell proliferation, migration, and invasion. Cancer Sci 2015, 106:1313-1322.
10. Thiaville MM, Stoeck A, Chen L, Wu RC, Magnani L, Oidtman J, Shih IM, Lupien M, Wang TL:Identification of PBX1 Target Genes in Cancer Cells by Global Mapping of PBX1 Binding Sites.Plos One 2012, 7.
11. Park JT, Shih Ie M, Wang TL: Identification of Pbx1, a potential oncogene, as a Notch3 target gene in ovarian cancer. Cancer Res 2008, 68:8852-8860.
12. Qiu Y, Tomita Y, Zhang B, Nakamichi I, Morii E, Aozasa K: Pre-B-cell leukemia transcription factor 1 regulates expression of valosin-containing protein, a gene involved in cancer growth. Am J Pathol 2007, 170:152-159.
13. Kim SK, Selleri L, Lee JS, Zhang AY, Gu XY, Jacobs Y, Cleary ML: Pbx1 inactivation disrupts pancreas development and in Ipf1-deficient mice promotes diabetes mellitus. Nature Genetics 2002,30:430-435.
14. Koss M, Bolze A, Brendolan A, Saggese M, Capellini TD, Bojilova E, Boisson B, Prall OWJ, Elliott DA, Solloway M, et al: Congenital Asplenia in Mice and Humans with Mutations in a Pbx/Nkx2-5/p15 Module. Developmental Cell 2012, 22:913-926.
摘要:
Background
Understanding the embryonic stem cell (ESC) fate decision between self-renewal and proper differentiation is important for developmental biology and regenerative medicine. Attention has focused on mechanisms involving histone modifications, alternative pre-messenger RNA splicing, and cell-cycle progression. However, their intricate interrelations and joint contributions to ESC fate decision remain unclear.
Results
We analyze the transcriptomes and epigenomes of human ESC and five types of differentiated cells. We identify thousands of alternatively spliced exons and reveal their development and lineage-dependent characterizations. Several histone modifications show dynamic changes in alternatively spliced exons and three are strongly associated with 52.8% of alternative splicing events upon hESC differentiation. The histone modification-associated alternatively spliced genes predominantly function in G2/M phases and ATM/ATR-mediated DNA damage response pathway for cell differentiation, whereas other alternatively spliced genes are enriched in the G1 phase and pathways for self-renewal. These results imply a potential epigenetic mechanism by which some histone modifications contribute to ESC fate decision through the regulation of alternative splicing in specific pathways and cell-cycle genes. Supported by experimental validations and extended datasets from Roadmap/ENCODE projects, we exemplify this mechanism by a cell-cycle-related transcription factor, PBX1, which regulates the pluripotency regulatory network by binding to NANOG. We suggest that the isoform switch from PBX1a to PBX1b links H3K36me3 to hESC fate determination through the PSIP1/SRSF1 adaptor, which results in the exon skipping of PBX1.
Conclusion
We reveal the mechanism by which alternative splicing links histone modifications to stem cell fate decision.
阅读论文全文请访问:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-018-1512-3?utm_source=WeChat&utm_medium=Website_linksSocial_media_organic&utm_content=CelZha-MixedBrand-multijournal-Multidisciplinary-China&utm_campaign=ORG_AWA_CZH_BMCWechat_dailyposts_blogs
期刊介绍:
Genome Biology (https://genomebiology.biomedcentral.com/) publishes outstanding research in all areas of biology and biomedicine studied from a genomic and post-genomic perspective.
The current impact factor is 13.214* and the journal is ranked 4th among research journals in the Genetics and Heredity category by Thomson Reuters. Genome Biology is the highest ranked open access journal in the category.
(来源:科学网)
特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。