|
|
|
|
|
MDPI Micromachines | 一种新颖的可编程自组装模块/纳米分子机器人: 具有柔性关节的DNA环基序列 |
|
|
论文标题:DNA Ring Motif with Flexible Joints (具有柔性关节的DNA环基序)
期刊:Micromachines
作者:Shiyun Liu, Satoshi Murata and Ibuki Kawamata
发表时间:31 October 2020
DOI:10.3390/mi11110987
微信链接:
https://mp.weixin.qq.com/s?__biz=MzI1MzEzNjgxMQ==&mid=
2649982783&idx=1&sn=8169ba2a52b11c51f7ddbfc8a4a6e3b5&
chksm=f1de3dfbc6a9b4edd9a5abfc64e2c56676e0c748c664fc812
bb906d71b5e79779cb29c04d8f5&token=227403453&lang=zh_CN#rd
期刊链接:
https://www.mdpi.com/journal/micromachines
原文作者简介

刘诗韵 (Shiyun Liu),日本东北大学工学研究科机器人学专业在读博士研究生。日本生物物理学会学生会员,日本计测控制学会学生会员,日本东北大学校长特别嘉奖留学生。主要从事DNA纳米技术,分子机器人学相关研究。致力于利用DNA折纸技术,开发大规模分子自组装DNA环基序列。

村田智 (Satoshi Murata),日本名古屋大学工学博士,日本东北大学工学研究科机器人学专业教授,博士生导师。日本生物物理学会会员,日本机械学会会员,日本计测控制学会会员,日本分子机器人学研究会主审。主要从事智能机器人学、知觉情报处理、自组装机器人学相关研究。主持日本文部科学省科研费"分子机器人学","分子控制论"等多项课题,发表SCI收录论文百余篇,授权国家发明专利3项。

川又生吹 (Ibuki Kawamata),日本东京大学情报理工学博士,日本东北大学工学研究科机器人学专业兼日本御茶水女子大学自然科学基础研究院讲师,日本分子机器人学研究会干事。主要从事DNA纳米技术、DNA分子计算、分子机器人学、自然语言处理相关研究。建立了面向DNA折纸超分子的合理设计平台,开发了DNA人工序列设计等数款软件。主持承担日本文部科学省科研费"分子机器人学","分子控制论"等多项课题,发表SCI收录论文数十篇。
制造具有期望形态的纳米尺度物体对于功能性分子装置的开发很重要,其中分子自组装技术作为组装具有所需形状和高精度纳米结构的DNA纳米技术,受到了极大的关注。DNA自组装主要有两种方法:DNA Tile和DNA折纸。DNA Tile是一种通过使用具有定义连通性的均质组件自组装晶格状纳米结构的方法,它能够以相对较少的碱基序列来构建大型2D结构,但是该结构的整体形状难以控制。DNA折纸是一种能够通过将长支架链与许多短钉链折叠来自动组装复杂形状的方法。尽管需要大量合成钉链,但我们可以通过DNA折纸组装2D和3D对象,它是结构DNA纳米技术中最强大,也是用途最广泛的方法。

来自日本东北大学的川又生吹教授及其团队在Micromachines期刊上发表了一篇题为“DNA Ring Motif with Flexible Joints”的文章。文中基于DNA折纸技术设计了一种新型的纳米级模块——柔性多关节环基序列,其形态、连接性和灵活性可以通过多种方式进行编程。该基序列可以通过其片段上的自我互补序列相互连接,且可以根据需要将柔性接头固定在伸直的位置,从而使序列呈现各种形态。同时可以通过调整柔性接头的数量和可连接片段的数量,从而实现序列的可编程自组装。作者成功制作了具有该特性的基序列并评估了几种自组装模型,该多关节环基序列为创建功能性分子装置提供了一种新的思路和方法。
实验设计
作者提出的DNA环基序列是由几个片段组成的DNA折纸多边形。由于支架链长度和制造序列片段受最小长度的限制,设计DNA环基序列为等边七边形。该七边形是最小的能够转化为两个不同的三角形的等边多边形,该方式为形状的可编程性提供了FC碰碰胡老虎机法典-提高赢钱机率的下注技巧的可能。
基序列中的一个片段由12个DNA双螺旋束组成,且其中10个双螺旋束以2 × 5晶格方式对齐排列成为序列片段的一部分,剩下的两个双螺旋束用于链接片段以构建完整的基序列 (图 1a)。每个片段中有13个连接线束,这些片段可以选择为可连接状态或不可连接状态。在可连接状态下,连接线束的顺序为自动互补的,允许片段之间相互连接,且连接线束在片段的外侧对称对齐排列,这样片段可以在基序列被翻转的状态下相互连接。为了避免片段之间的错位,将排列在片段中心的连接线束设计为具有最强结合能力,而四角的连接线束被设计成具有最弱结合能力。在不可连接状态下,可以通过使用多T序列而不是自互补序列来防止片段之间的连接 (图1b)。

图1.具有柔性关节的DNA环基序列的设计概要。(a) 等边七边形DNA环基序列的主要构成;(b) 可连接状态与不可连接状的片段;(c) 柔性关节的固定与解除;(d) 不同形状的基序列的自组装示意图。
这些片段由两个单链DNA连接,这是支架链的一部分。由于单链DNA比DNA双螺旋具有更高的灵活性,这两条单链DNA链可以充当一个柔性关节,从而使基序列呈现不同的形状。通过添加12个单链DNA,可以直接固定基序列的关节 (图 1c)。
通过固定接头和选择不同位置段的状态,可以编程基序列的形状和连通性。利用基序形状和连通性的可编程性,可以实现各种自组装模型 (图 1d)。
基序列的形状和连通性可以通过固定关节并选择不同位置的片段状态来编程。利用基序列形状和连通性的可编程性,可以实现各种自组装模型。
实验结果
1. 形状控制
在实验中,作者观察到了七边形和两个不同三角形形状的基序列。在这个过程中,由于三角形的形状稳定,易于观察和分析,作者将其选为观察目标。通过测量三角形顶点的数量、边长、内角和序列的周长来判断基序列的几何特征。结果表明,顶点数量分布在3–7范围内的不同值中。此外,图2a,b中内角的分布和边长表明,基序列呈现出不同的形状,这证明了由自由关节引起的柔韧性。通过两种不同的方式固定基序列的四个关节,作者制作了 (2, 2, 3) 和 (3, 3, 1) 两种等腰三角形,其中 (2, 2, 3) 表示等腰三角形三个边分别由2个片段,2个片段和3片个段组成。实验中,作者成功观测到了两种形状不同的三角形。图2c-f 显示内角的分布和两个顶点之间的距离出现两个峰值,表明该基序列按照设计变为等腰三角形的形状。
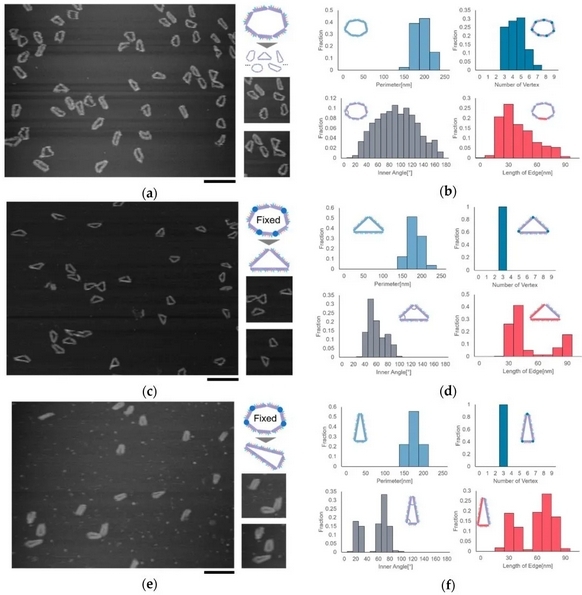
图2.具有柔性关节的DNA环基序列的制作与观察。(a, b) 等边七边形基序列的AFM观察与解析结果;(c-f) 由等边七边形变形而来的两种等腰三角形基序列的AFM观察与解析结果。
2. 自组装结构
作者通过不同形状和连接变换组合观察基序列的自组装 (图3)。结果显示,仅含有一个可连接链片段的七边形基序列形成二聚体,且二聚体的产率超过80%。最短边 (一个片段) 处于可连接状态的三角形 (3, 3, 1) 基序也形成了二聚体,但其二聚体的形状不同于由七边形基序形成的二聚体。最长边 (3个片段) 处于可连接状态的三角形 (2, 2, 3) 基序大部分形成二聚体。由于自互补序列的集合是相同的,当片段在移动的位置连接其他序列时,基序列可以自组装成线性多聚体。

图3.具有柔性关节的DNA环基序列自组装。(a) 由等边七边形所形成的二聚体的观察与解析结果;(b) 由等腰三角形 (3, 3, 1) 所形成的二聚体的观察与解析结果;(c) 由等腰三角形 (2, 2, 3) 所形成的二聚体与线性多聚体的观察与解析结果。
结论
在这项研究中,作者提出了一种具有形状、连接性和灵活性可编程的自组装模型,成功地制作了目标基序列并将其组装成不同的结构。该自组装原理新颖简单,以可编程方式形成自组装结构,为将来在更大范围内实现具有更高复杂性的自组装提供了参考,且该研究进一步丰富了分子机器人学理论在DNA纳米技术中的应用。
该文入选Micromachines期刊第11卷11期封面文章,博士研究生刘诗韵为第一作者,川又生吹博士为本文通讯作者。
Micromachines (ISSN 2072-666X; IF 2.523) 是一个与微纳米技术领域相关的国际型开放获取期刊,期刊范围涵盖微纳米结构、材料、设备和系统等各方面的研究与应用。目前期刊已被SCIE (Web of Science),Ei Compendex,Scopus等数据库收录。Micromachines采取单盲同行评审,一审周期约为12.5天,文章从接收到发表仅需1.9天。


特别声明:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用,须保留本网站注明的“来源”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜,请与我们接洽。